
该研究题为《Development of machine learning prediction models for systemic inflammatory response following controlled exposure to a live attenuated influenza vaccine in healthy adults using multimodal wearable biosensors in Canada: a single-centre, prospective controlled trial》由Amir Hadid、Emily G McDonald、Qianggang Ding、Christopher Phillipp、Audrey Trottier、Philippe C Dixon、Oussama Jlassi、Matthew P Cheng、Jesse Papenburg、Michael Libman和Dennis Jensen等撰写,作者隶属于加拿大蒙特利尔麦吉尔大学教育学院运动机能学与呼吸生理学实验室、麦吉尔大学Sylvan Adams运动科学研究所、麦吉尔大学健康中心内科总医学科、魁北克人工智能研究所(Mila)、蒙特利尔大学计算机科学与运筹学系以及麦吉尔大学健康中心研究所。研究旨在开发基于多模态可穿戴生物传感器与机器学习的预测模型,用于实现健康成年人在接种减毒流感活疫苗(LAIV)后系统性炎症反应的早期预测,而无需依赖临床症状。该团队在加拿大蒙特利尔麦吉尔大学健康中心招募了56名18–59岁的健康志愿者,通过Oura指环、Biobeat手表和Astroskin–Hexoskin智能衬衫等设备持续监测心率、呼吸频率、加速度等生理数据,并结合高时间分辨率的血液炎症生物标志物进行分析。采用梯度提升机器学习算法(XGBoost)训练模型后,在实时预测和24小时容差预测窗口下的ROC-AUC分别达到0.73和0.89,显著优于仅基于症状的检测模型(ROC-AUC分别为0.66和0.79)。研究结果表明,融合系统性炎症生物标志物与可穿戴设备生理数据的机器学习模型,可实现对轻微流感感染引发的炎症反应的早期客观检测,有望推动病毒性呼吸道感染(包括无症状或亚临床阶段)的早期发现,为公共卫生干预和防控策略提供新的技术手段。
01 摘要
背景:与基于症状的检测相比,症状前或无症状的免疫系统信号以及亚临床生理变化或许能更客观地衡量早期病毒性上呼吸道感染 (VRTI)。我们旨在利用多模态可穿戴传感器、宿主反应生物标志物和机器学习技术,在不依赖症状的情况下,预测受控接种减毒活流感疫苗后的全身性炎症。
方法:WE SENSE 研究是一项单中心(加拿大魁北克省蒙特利尔市麦吉尔大学健康中心)前瞻性对照试验,招募了 18-59 岁的健康成年人,他们在研究期间未接种或不计划接种季节性流感疫苗或任何其他疫苗。我们排除了筛选前 7 天内有任何传染症状的参与者。我们通过使用智能戒指(Oura ring Gen 2,芬兰 Oura Oy)、智能手表(Biobeat watch,以色列 Biobeat Technologies)和智能衬衫(Astroskin–Hexoskin shirt,加拿大 Hexoskin)进行持续监测收集生理和活动数据(例如心率、呼吸频率和加速度),以及 12 天内(接种前 7 天和接种后 5 天)的高时间分辨率全身炎症生物标志物图谱。我们经常在接种前后通过 PCR 检测参与者的呼吸道病原体,并通过应用程序监测他们的症状和自由文本注释。使用梯度提升技术对预测全身炎症激增的机器学习算法进行了训练(35 名参与者)、验证(10 名参与者)和测试(10 名参与者)。
发现:2021年12月10日至2022年2月28日期间,我们招募了56名参与者,其中55名有可用数据;所有55名参与者均持续佩戴Oura戒指,54名参与者穿着Astroskin-Hexoskin衬衫,50名参与者佩戴Biobeat手表。27名(49%)参与者为女性,28名(51%)为男性;31名(56%)参与者为白人,8名(15%)为亚裔,4名(7%)为黑人,2名(4%)为拉丁裔或西班牙裔,10名(18%)未透露种族。我们使用模型2作为候选模型,该模型包含从Oura戒指夜间数据中精心挑选的特征,因为它基于最少的特征数量构建(更实用)。该模型使用 Oura 环的夜间数据,预测炎症激增的受试者工作特征曲线下面积 (ROC-AUC) 为实时预测 0.73(95% CI 0.71–0.74),24 小时耐受性预测窗口 (24h-tol) 0.89(0.87–0.90)。结合 Astroskin–Hexoskin 衬衫的夜间和日间数据,实时 ROC-AUC 值为 0.73(0.71–0.75),24 小时耐受性预测窗口为 0.91(0.90–0.92),且精确度有所提高(即特异度 [0.83, 0.79–0.87] 和 F1 得分 [0.65, 0.58–0.71])。仅基于症状的模型性能较低,实时 ROC-AUC 值为 0.66(0.63–0.68),24 小时-tol 为 0.79(0.77–0.82)。
解释:全身炎症生物标志物与可穿戴生物传感器的生理数据相结合,提供了丰富客观的数据,可用于训练机器学习算法,预测低度流感病毒引起的全身炎症。这种方法优于基于症状的检测,并有可能提高对流感等VRTI的检测水平,并缩短检测时间,即使在无症状人群中也是如此。
资助:本研究由加拿大健康研究院(Canadian Institutes of Health Research)资助。
02 研究背景
病毒性上呼吸道感染(VRTIs)是全球范围内常见且影响广泛的公共卫生问题,传统的症状监测方法存在检测延迟和主观偏差,难以及时发现无症状或亚临床感染个体。这种延迟不仅限制了个体的早期治疗窗口,还增加了疾病传播的风险。随着可穿戴设备在生理监测中的广泛应用,研究者开始探索如何通过可穿戴传感器实时捕获心率、呼吸频率、体温等生理信号,以识别潜在的炎症反应。然而,目前缺乏系统性研究将可穿戴设备数据与免疫生物标志物结合,并基于机器学习构建预测模型,实现对感染早期炎症反应的敏感检测。
二、研究难点
(1)早期检测的敏感性与特异性:可穿戴设备采集的生理信号存在噪声和个体差异,如何提取出与炎症反应高度相关的特征并避免误判(如运动、情绪波动等引起的生理变化)是挑战之一。
(2)亚临床感染的检测:部分感染个体在早期阶段无明显症状,仅凭症状难以发现,如何利用连续监测数据及微弱的免疫信号提前预警,是实现“无症状人群早期识别”的关键技术难题。
(3)多模态数据融合:将可穿戴设备多模态数据(如心率、呼吸、活动强度)与血液炎症生物标志物有效融合,并通过机器学习构建高效预测模型,需要解决异质数据标准化、特征选择和模型泛化能力等技术瓶颈。
三、相关工作
过去十年,尤其是COVID-19大流行期间,可穿戴设备在感染早期检测中的应用研究不断增加。一些研究发现,心率变异性(HRV)的变化可作为系统性炎症反应的潜在指标。例如,有研究报道在COVID-19患者中,HRV的显著下降与C-反应蛋白(CRP)的升高相关联。然而,现有研究多数停留在单一生理参数监测层面,缺乏结合炎症生物标志物的系统性分析,也未形成在真实世界环境下验证的预测模型。此外,虽然已有机器学习和AI算法尝试基于可穿戴设备数据检测SARS-CoV-2感染,但大多依赖症状记录或病毒载量检测,未能突破亚临床阶段的检测难题。针对这一空白,本研究提出融合可穿戴设备多模态生理数据与高时间分辨率炎症生物标志物,开发机器学习预测模型,以实现健康成年人在低剂量病毒暴露(减毒流感活疫苗接种)后的系统性炎症早期检测。
03 模型设计
数据采集与处理
研究在健康成年人中开展,受试者连续佩戴多模态可穿戴设备,包括Oura指环(监测心率、心率变异性、体温和睡眠)、Biobeat手表(监测血压、血氧饱和度、呼吸频率)、Astroskin–Hexoskin智能衬衫(记录心电图、连续呼吸和加速度数据)。
同时高频采集血液样本,检测48种系统性炎症生物标志物,计算炎症折叠变化(IFC)并进行归一化处理(nIFC),用于标注炎症反应状态(是否存在炎症)。
对可穿戴设备数据进行清洗,剔除生理范围外的异常值,确保接种前后数据完整性和质量(覆盖率超过50%)。
特征工程
根据Oura指环记录的睡眠时间,将数据分为夜间和白天两部分,以减少活动状态干扰。
提取统计特征,包括均值、最大值、最小值、趋势(线性斜率)、标准差等,构建特征矩阵。
对所有特征采用Z-score标准化,减少不同个体间的差异,并整合锻炼、咖啡和酒精摄入等潜在混杂因素。
模型构建与训练
采用梯度提升决策树(XGBoost)作为核心算法,针对二分类任务(炎症反应/无炎症)训练预测模型。
将55名参与者随机分为训练集(35人)、验证集(10人)、测试集(10人),其中4名接种后意外感染SARS-CoV-2的参与者作为外部验证组。
设计8种不同组合的模型,包括基于Oura指环夜间数据、Astroskin–Hexoskin夜间和白天数据、Biobeat夜间数据及症状数据的模型,进行性能比较和优化。
性能评估与优化
使用ROC-AUC、Precision-Recall-AUC、敏感性(Sensitivity)、特异性(Specificity)和F1分数等指标评估模型表现。
在保持高敏感性(约90%)前提下优化分类阈值,实现早期检测需求。
最终确定基于Oura指环夜间数据的模型(模型2)为候选模型,其实时预测和24小时容差预测的ROC-AUC分别为0.73和0.89。
04 实验设置
1. 数据集
本研究在加拿大蒙特利尔麦吉尔大学健康中心开展,招募了56名18–59岁的健康成年人,其中55人数据完整。所有参与者在研究期间未接种流感疫苗或其他疫苗,且筛查前7天内无感染症状。
研究周期共12天,包括接种前7天(基线期)和接种后5天(随访期)。
数据来源包括:
可穿戴设备连续监测数据:Oura指环(心率、心率变异性、体温、活动、睡眠)、Biobeat手表(血压、血氧、呼吸率)、Astroskin–Hexoskin衬衫(心电图、呼吸、加速度)。
炎症生物标志物:血液样本检测48种炎症因子,计算归一化炎症折叠变化评分(nIFC)作为模型标签(是否发生炎症)。
PCR检测与症状自报数据:用于分析感染状态和验证模型预测效果。
数据集划分:训练集(35人)、验证集(10人)、测试集(10人),另有4名接种后感染SARS-CoV-2的参与者作为外部测试组。
2. 基线方法
研究设计并比较了8种模型:
多模态可穿戴设备模型:基于Oura指环、Biobeat手表、Astroskin–Hexoskin衬衫的不同数据组合(夜间/白天、自动特征选择/手工特征选择)。
症状模型:基于Wisconsin上呼吸道症状调查表(WURSS-21)自报症状构建的预测模型,作为传统方法基线。
组合模型:可穿戴设备数据与症状数据联合输入模型,探索多信息融合的效果。
所有模型均采用XGBoost梯度提升决策树作为核心算法,通过5折交叉验证优化参数。
3. 评估指标
主要评估指标:
ROC-AUC(受试者工作特征曲线下面积):衡量模型整体分类性能。
Precision-Recall-AUC(精确率-召回率曲线下面积):评估模型在不平衡数据集上的表现。
次要评估指标:
敏感性(Sensitivity):检测炎症事件的能力。
特异性(Specificity):排除非炎症事件的能力。
F1分数:平衡精确率与召回率,反映模型在正负样本上的综合表现。
实时预测与24小时容差预测两种场景下分别计算上述指标,以验证模型在不同时间窗口内的适用性。
05 结果与分析
数据概况
炎症反应检测
模型性能
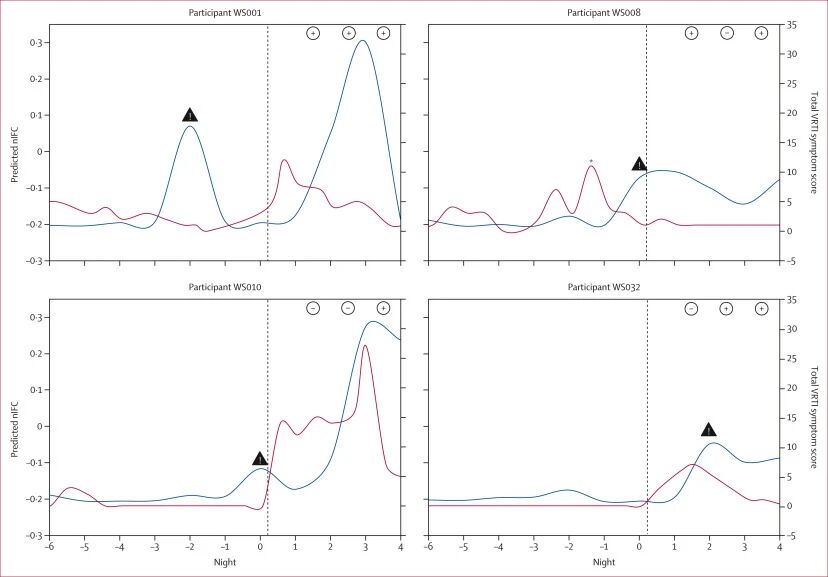
候选模型(基于Oura指环夜间数据)在实时预测和24小时容差预测中的ROC-AUC分别达到0.73(95% CI: 0.71–0.74)和0.89(95% CI: 0.87–0.90)。
在加入Astroskin–Hexoskin夜间和白天数据后,模型性能进一步提升,ROC-AUC分别为0.73(95% CI: 0.71–0.75)和0.91(95% CI: 0.90–0.92),并且在特异性(0.83)和F1分数(0.65)上均优于单纯基于症状的模型。
对比基于症状的模型,其ROC-AUC仅为0.66(实时预测)和0.79(24小时容差预测),性能明显较低。
重要特征贡献
06 结论
本研究成功开发并验证了基于多模态可穿戴生物传感器数据和系统性炎症生物标志物的机器学习预测模型,实现了对健康成年人在接种减毒流感活疫苗(LAIV)后系统性炎症反应的早期预测。研究结果表明,融合可穿戴设备连续监测的心率、呼吸频率、体温等生理信号与高时间分辨率炎症指标,可以在无症状或亚临床阶段敏感捕捉炎症变化。相比单纯基于症状的传统方法,所构建的模型在实时预测和24小时容差预测中的ROC-AUC分别达到0.73和0.89,显著提升了检测性能。
该研究证明了可穿戴设备与机器学习结合的潜力,有望为病毒性呼吸道感染(如流感、SARS-CoV-2等)的早期识别提供新工具,推动个体化健康管理和公共卫生防控策略的智能化发展。未来,该方法可拓展应用于其他炎症相关疾病的监测与干预,具有广阔的临床与公共卫生应用前景。
07 论文评价
✅创新亮点
融合多模态可穿戴设备数据与炎症生物标志物
亚临床和无症状炎症反应的敏感检测
高性能机器学习模型开发
真实世界可应用性强
面向未来的临床和公共卫生价值
⚠方法不足
本研究存在一定局限性。首先,样本量较小且仅包含18–59岁的健康成年人,缺乏老年人、儿童及慢性病患者数据,限制了模型的泛化能力。其次,研究监测周期仅12天,未能验证模型在长期连续监测下的稳定性。此外,实验未采用双盲设计,受试者知晓已接种疫苗可能引发安慰剂效应或症状过度报告,影响症状数据的真实性。虽然模型融合了多模态生理数据和炎症生物标志物,但部分标志物在个体间响应差异较大,存在潜在偏倚风险。模型阈值设定偏向高敏感性以确保早期检测,但特异性相对较低,可能导致中度炎症水平时出现误报。最后,方法高度依赖可穿戴设备的连续数据,现实环境中用户依从性和设备稳定性可能影响预测效果。
08 参考资料
论文链接:
10.1016/j.landig.2025.100886